
Masu
こんにちは、製薬研究員のMasuです。
遺伝子やタンパク質の網羅的解析を行ったら、それらの変動がどんな意味を持つか調べたいですよね?
このページでは、KEGG pathway解析をDAVIDというサイトを利用して行う方法を紹介します。
DAVID
DAVIDでは、解析を行った遺伝子もしくはタンパク質のIDを用いてKEGG pathwayKEGG pathway解析やGO (Gene Ontology) 解析を行うことが出来ます。
まずこれらの解析を行うには、実際に解析を行った遺伝子もしくはタンパク質のIDをDAVID上にアップロードする必要があります。
①まずFunctional Annotationを開く(写真赤枠)
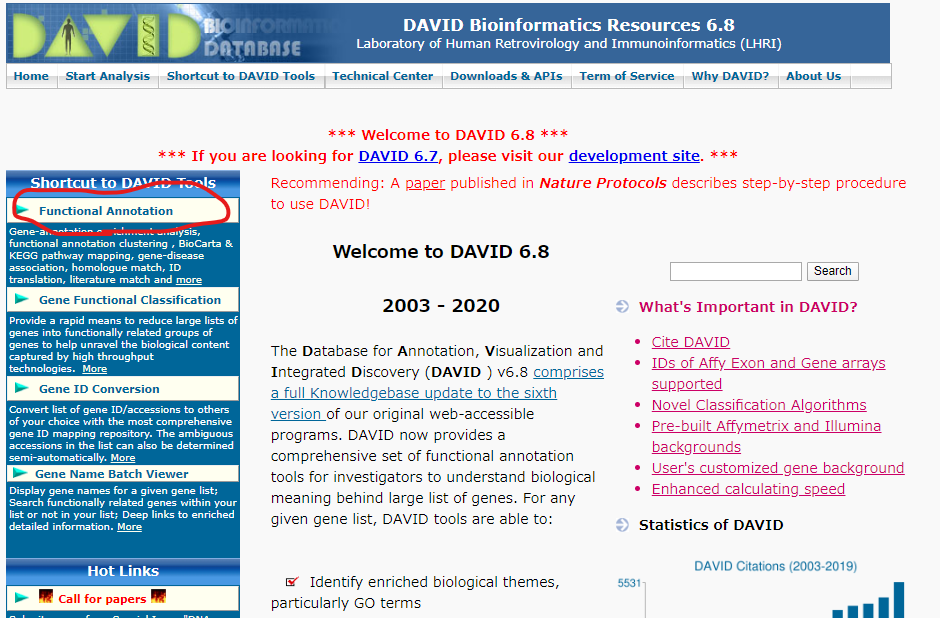
②左のEnter Gene Listの所に解析した遺伝子もしくはタンパク質のIDリストを張り付けてください

③Select Identifierの所でIDの名称を選択します(例:P08684(human CYP3A4) → UNIPROT_ACCESSION)

④Gene Listを選択し、submitします。

⑤すると以下のような画面が出てきますので、KEGG_PATHWAYのタブを開き、赤枠で示したChartボタンを選択します。
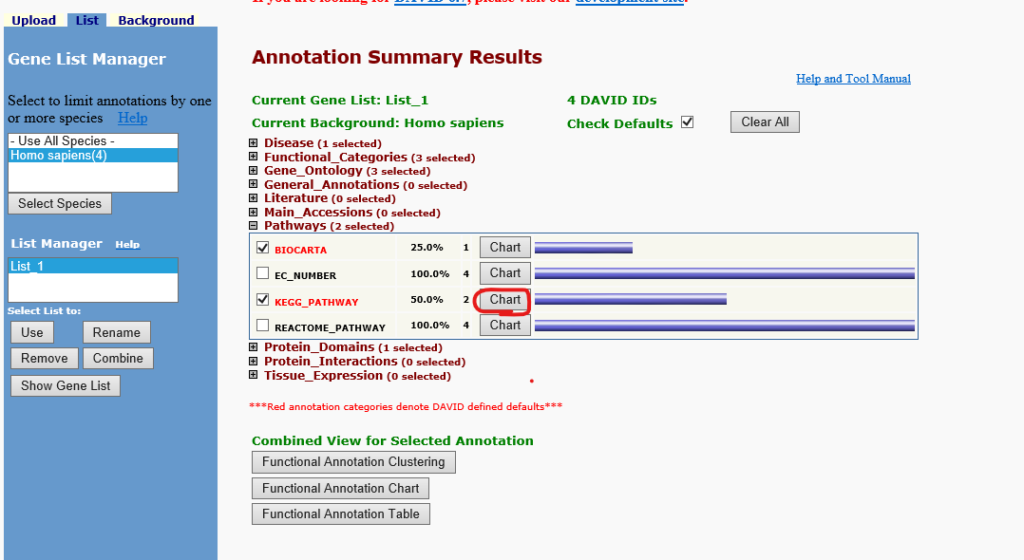
これで完了です。これらの操作によって、以下のような画面が出てくると思います(今回は4つしか遺伝子名を入れていないので、表示されるpathwayの数が少なくなっています)。
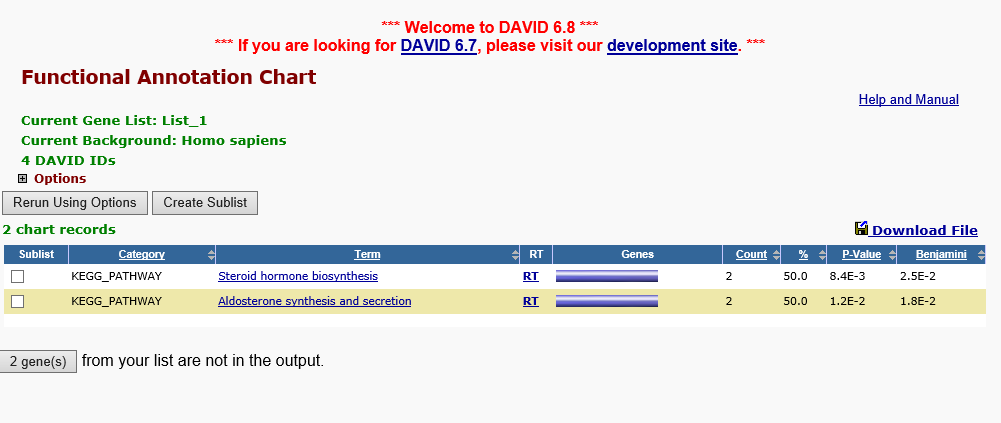
Termと記載されているものがpathwayの名前です。上から関連性が高い順に並んでいます。
あとは気になったpathwayを選択することで、より詳細な解析が行えると思います。



コメント